上古神农氏尝百草以明药性,今天人工智能也能在数字世界中“尝试”药物,预测它们对不同类型肿瘤细胞的作用效果。
近日,良渚实验室/浙江大学基础医学院郭国骥、韩晓平、夏宏光等团队合作推出了一个创新的深度学习框架“神农”(Shennong),为筛选抗肿瘤药物提供了新途径。相关研究发表在《National Science Review》。
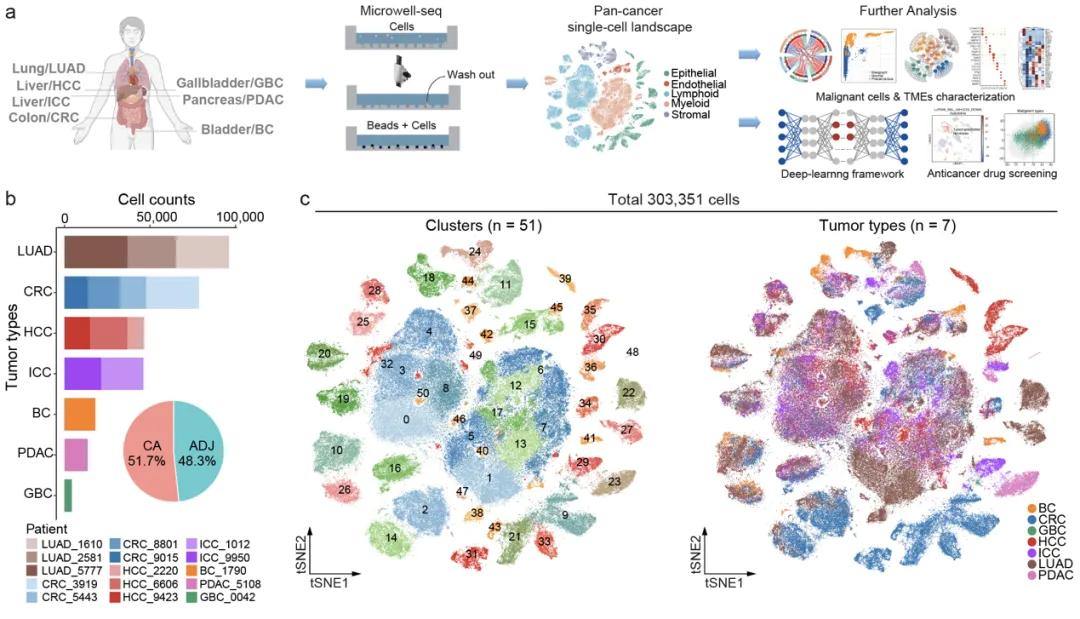
目前,抗肿瘤药物筛选主要依靠转录组测序数据分析,从而了解一大群肿瘤细胞的基因信息,判断药物效果。但这种方法往往忽视了肿瘤内部具有复杂性,每个细胞都可能不一样,而且肿瘤细胞和它周围环境也会互相影响。另外,很多研究只关注单一癌症类型,没有充分利用不同癌症之间可能有的共同点。
因此,亟需开发基于单细胞泛肿瘤图谱的药物筛选方法,这个图谱就像一张详细的“细胞地图”,记录了各类肿瘤中每个细胞的特征。通过分析这些细胞数据,研究人员就能在微观层面上,更有效地筛选抗肿瘤药,并且能更清楚地了解这些药物的作用机制和潜在的副作用。
传统的药物筛选或药物发现方法通常耗时且成本高昂。人工智能和机器学习方法加速了药物发现和开发的进程。研究人员推出了神农框架,正是这类方法的创新典范。
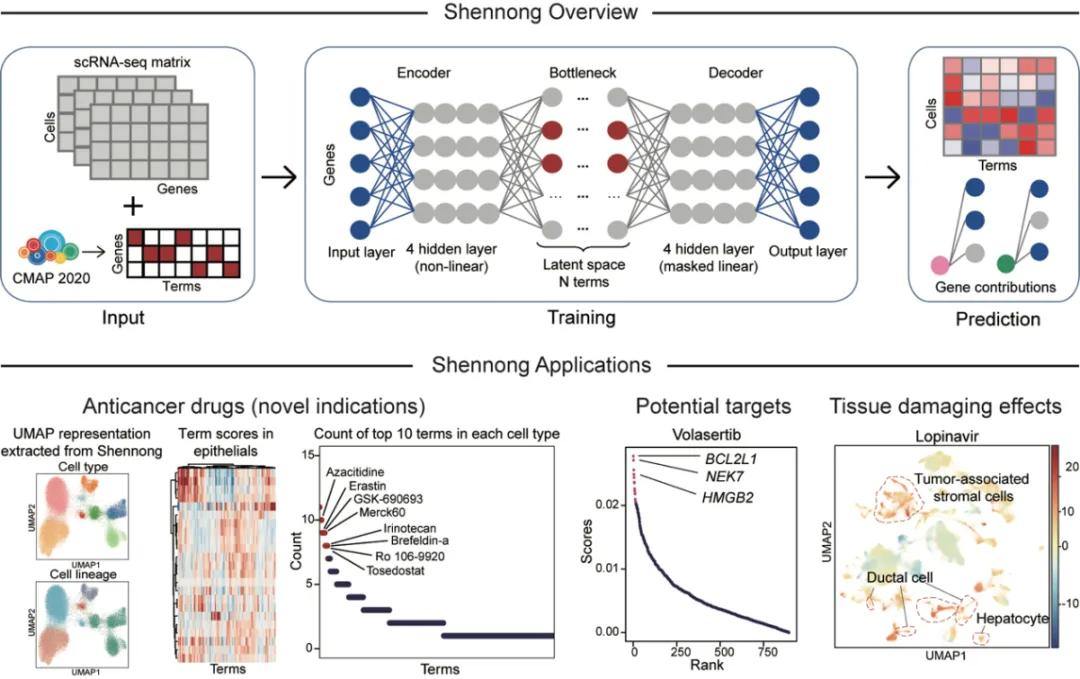
神农框架能同时观察数以万计的细胞在接触药物后的反应。不同于简单的“有效/无效”判断,它会从多个维度来理解和描述每个细胞的变化。分析系统采用深度学习模型,就像一位能够快速处理海量信息的“药物侦探”。
它不仅能提取细胞反应中最关键的特征,还能通过概率的方式来描述这些变化:不是简单地说“这个药物对这类细胞有效”,而是能够详细描述“这个药物有多大可能性、以什么方式影响了细胞”。这种细致入微的分析方法,让研究人员能更全面地理解药物的作用机制,甚至发现药物的新用途。
将神农框架应用于泛肿瘤细胞图谱后,它不仅能预测药物的效果和副作用,帮助筛选药物,还能发现一些已经批准的药物,它们可能对其他癌症也有用。
例如,乳腺癌治疗药物“依西美坦”,研究发现对肺癌细胞表现出显著的抗增殖效果,同时这一发现在一期临床试验中得到证实。
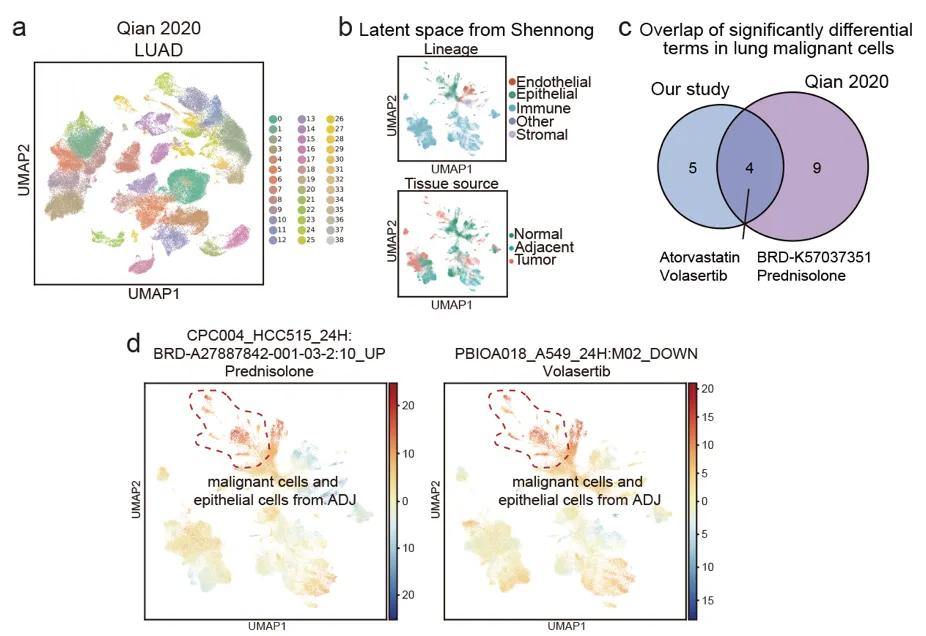
为了验证神农框架的准确性,研究人员还把它的预测结果和其他第三方数据集的结果进行比较。结果发现,框架能够从不同来源的数据中提取出细胞的独特和共性特征,而且预测结果有30%到45%的重叠,这表明,神农框架具有良好的稳健性和泛化能力。
“神农框架展现出良好的稳健性和可解释性,有望显著提升药物筛选的效率和准确度,加速新药开发进程。”良渚实验室团队相关负责人表示。
记者 赵艺乔 通讯员 李弘慧



